visualization of regression models
회귀모형
사실 많은 사람들이 회귀모형이라고 하면 선형회귀를 생각하지만, 회귀 모형은 결과 변수가 연속형인 경우에 쓰이는 일반(?) 명사이다. (보통 예측 모형을 회귀와 분류로 나누지 않는가?)
회귀모형은 선형회귀모형처럼 비교적 이해하기 쉬운 모형부터 인공신경망처럼 흔히 Black-Box 모형이라고 불리는 복잡한 모형까지 모두 포함된다.
visreg : visualization of regression models
데이터에 적합된 회귀 모형을 좀 더 이해하기 위해 사용할 수 있는 여러 방법에 대해 이미 몇 가지 방법을 알아보았다. 각 패키지들은 나름의 장점과 단점을 가지고 있었다(예. 속도의 차이, 잔차를 그려주는가? 등)
visreg는 앞서 소개한 패키지와 능히 견줄만한 패키지로 다양한 모형에 대해 시각화를 도와 준다. 예를 들어 예측 함수를 그려보고, 잔차도 그려보고, 조건부 그림도 그려볼 수 있다.
데이터
앞에서 소개했던 인공 데이터를 다시 활용해보자.
library(dplyr)
library(ggplot2)
TURE <- TRUE
genData = function(N=1000, iseed=100) {
set.seed(iseed)
sdErr = 1
x1 <- runif(N, -3, 3)
x11 <- x1^2
x2 <- runif(N, -3, 3)
x3 <- sample(c(-1, 1), N, replace=TRUE)
ncolMatX <- 5
ncolMatX2 <- 2
require(mvtnorm)
sigma <- matrix(
rep(0.97, ncolMatX*ncolMatX),
ncolMatX, ncolMatX)
diag(sigma) = 1
matX <- rmvnorm(N, mean=rep(0,ncolMatX), sigma=sigma)
x40 <- apply(matX, 1, mean)/ncolMatX
colnames(matX) <- paste0('x', 4:(ncolMatX+3))
matX2 <- rmvnorm(N, mean=rep(0,ncolMatX2), sigma=diag(ncolMatX2))
colnames(matX2) <- paste0('x', (ncolMatX+4):(ncolMatX+ncolMatX2+3))
y <- x11 + x2/(abs(x1)+0.1) + (x2 < 0)*x40 + (x2<1)*x3 * x40 + rnorm(N, 0, sdErr)
return(data.frame(y, x1, x2, x3, matX, matX2))
}
dat <- genData()
모형 적합
fitLM <- lm(y ~ ., data=dat) ## linear model
library(randomForest)
fitRF <- randomForest(y ~ ., data = dat, importance = TRUE)
library(gbm) # install.packages('gbm')
fitGBM <- gbm(y ~ ., data=dat, dist="gaussian", inter=2, n.trees=1000)
library(gam) ## gam # install.packages('gam')
fitGAM <- gam(y ~ s(x1)+s(x2)+x3+s(x4)+s(x5)+s(x6)+s(x7)+s(x8)+s(x9)+s(x10),
data=dat) # x3 is a categorical variable
library(e1071)
fitSVM <- svm(y ~ ., data=dat)
library(nnet) ## nnet
set.seed(4)
fitNN10 <- nnet(y ~ ., data=dat,
linout=TRUE, size=10, decay=0.01, maxit=1000,
trace=FALSE)
#fitNN20 <- nnet(y ~ ., linout=TRUE, size=20, data=dat)
fitNN40 <- nnet(y ~ ., linout=TRUE, size=40, data=dat, maxit=1000, trace=FALSE)
예측 함수와 잔차
library(visreg)
visreg(fitRF, "x1", main='fitRF w.r.t x1')
visreg(fitRF, "x2", main='fitRF w.r.t x2')
visreg(fitGBM, "x1", main='fitGBM wrt x1')
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
visreg(fitGBM, "x2", main='fitGBM wrt x2')
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
visreg(fitGAM, "x1", main='fitGAM wrt x1')
visreg(fitGAM, "x2", main='fitGAM wrt x2')
visreg(fitSVM, "x1", main='fitSVM wrt x1')
visreg(fitSVM, "x2", main='fitSVM wrt x2')
visreg(fitNN10, "x1", main='fitNN10 wrt x1')
visreg(fitNN10, "x2", main='fitNN10 wrt x2')
visreg(fitNN40, "x1", main='fitNN40 wrt x1')
visreg(fitNN40, "x2", main='fitNN40 wrt x2')
여기서 각 점들은 예측값이 아니라 잔차를 시각화하였음에 유의하자. 모형에 따라 잔차가 어떻게 달라지는지 확인해 살펴보자. 예측 함수가 구불구불할 수록 잔차가 작아지는 경향이 있다.
gbm 처럼 predict 또는 residual 함수를 지원하지 않는 경우에는 간단하게 만들어 줄 수 있다.
residuals.gbm <- function(fit) {fit$data$y - fit$fit}
# https://pbreheny.github.io/visreg/blackbox.html
visreg(fitGBM, "x1")
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
visreg(fitGBM, "x2")
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
조건부 예측
visreg(fitRF, "x1", by='x2')
visreg(fitRF, "x1", by='x3')
visreg(fitRF, "x2", by='x1')
visreg(fitGBM, "x1", by='x2')
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
visreg(fitGBM, "x1", by='x2')
## Warning in plot.visreg(v, ...): The generic function residuals() is not set ## up for this type of model object. To plot partial residuals, you will need ## to define your own residuals.gbm() function.
visreg(fitGAM, "x1", by='x2')
visreg(fitSVM, "x1", by='x2')
visreg(fitNN10, "x1", by='x2')
visreg(fitNN40, "x1", by='x2')
추가 사항
rug를 넣어주거나, ggplot2를 사용하여 그림을 추가할 수도 있다.
visreg(fitRF, 'x1', rug=2, main='fitRF with rug')
visreg(fitRF, 'x1', gg=TRUE) +
labs(title='fitRF: response and residuals') +
geom_smooth(data=dat, aes(x=x1, y=y), col='green', alpha=0.4)
## `geom_smooth()` using method = 'gam' and formula 'y ~ s(x, bs = "cs")'
2d/3d 그림
두 변수에 대해 2D로 그리거나,
visreg2d(fitRF, 'x1', 'x2')
visreg2d(fitGAM, 'x1', 'x2')
visreg2d(fitNN10, 'x1', 'x2')
3D로 그릴 수도 있다.
visreg2d(fitRF, "x1", "x2", plot.type="rgl")

visreg2d(fitRF, "x1", "x2", plot.type="persp")
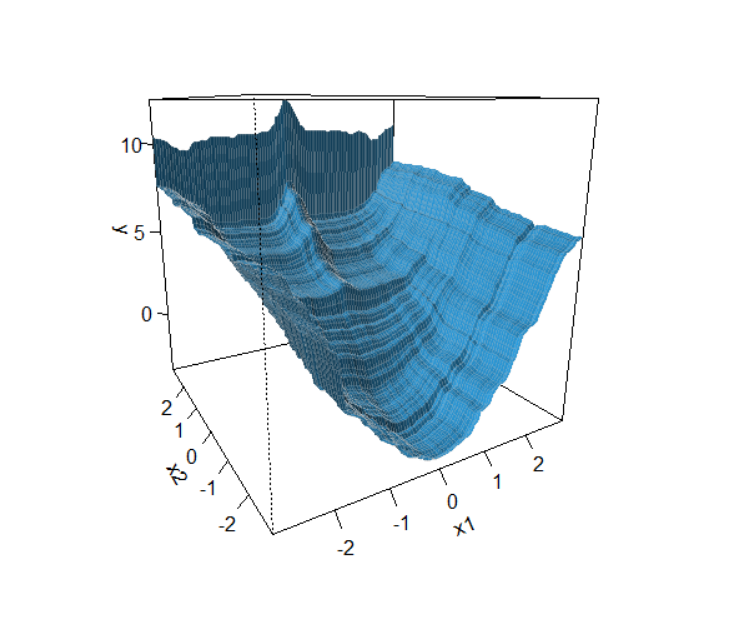
plot.type='persp'은 지나치게 오래 시간이 걸리는 게 흠이다.
그 밖에
그 밖에도 glm, coxph, rlm, locfit, quantreg 등을 지원한다고 한다.
Leave a comment